Équipe Philippe RONDARD
Neurorécepteurs, dynamique et fonctions

L’équipe a une longue expérience dans le domaine de la neuropharmacologie et l’étude des récepteurs couplés aux protéines G (RCPG), qui sont la cible de plus de 30% des médicaments. Notre équipe a réalisé d’importantes découvertes dans le domaine de la pharmacologie moléculaire de deux types de récepteurs, les récepteurs du glutamate (mGlu) et le récepteur GABA-B, qui sont la cible de neurotransmetteurs majeurs, le glutamate et le GABA, respectivement. L’étude de leurs mécanismes moléculaires par des techniques biophysiques et biochimiques innovantes a conduit à la découverte de nouveaux modulateurs allostériques avec des applications importantes pour les maladies neurologiques et psychiatriques.
Les récepteurs mGlu et GABA-B sont des récepteurs couplés aux protéines G complexes qui fonctionnent sous la forme d’homodimères ou d’hétérodimères. Ils sont également capables de former des complexes avec d’autres protéines, y compris des protéines membranaires, pour moduler leur activité in vivo. Il est important de mieux comprendre les bases moléculaires et structurales de ces complexes et leur dynamique, en particulier pour développer de nouvelles molécules d’intérêt thérapeutique dans le domaine des maladies neurologiques et psychiatriques.
Le projet de recherche de l’équipe a quatre objectifs spécifiques :
1 – Étudier les bases moléculaires et structurales des récepteurs mGlu et des complexes qu’ils forment avec d’autres partenaires membranaires, par des approches biophysiques et structurales (cryo-électron microscopie, analyse de la dynamique conformationnelle à l’échelle de la molécule unique par la technique de transfert d’énergie de résonance de type Förster ou FRET).
2- Développer des méthodes et des outils pour l’étude des récepteurs mGlu et GABA-B in vitro et dans leur environnement natif. Les méthodes privilégiées sont des biosenseurs basés sur les technologies de transfert d’énergie de résonance de type Förster (FRET, HTRF®) ou de bioluminescence (BRET). Les outils développés sont des ligands spécifiques, des petites molécules ou des anticorps de camélidés (nanobodies), et certains d’entre eux sont contrôlables par la lumière.
3- Étudier les complexes formés par les récepteurs mGlu et leur dynamique dans leur environnement natif. L’objectif est d’identifier des complexes fonctionnels dans le cerveau, tels que les récepteurs mGlu hétérodimériques que l’équipe a récemment découverts. Et de comprendre l’importance physiologique de ces complexes pour le fonctionnement des neurones et du cerveau. Les outils décrits ci-dessus sont utilisés.
4 – Développer les nanobodies ciblant les récepteurs mGlu comme immunothérapie innovante pour le traitement de maladies du cerveau, en particulier la schizophrénie.
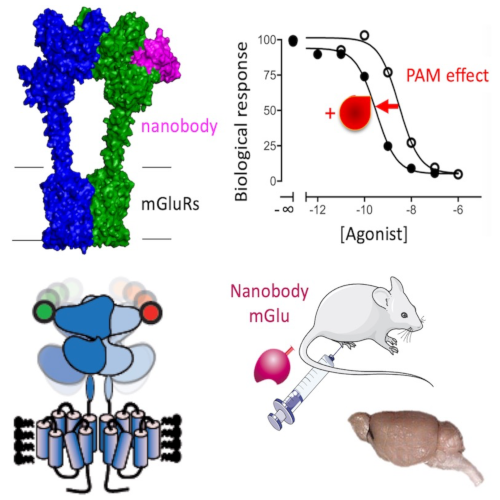
L’équipe étude la pharmacologie moléculaire des récepteurs mGlu et GABA-B, depuis leur structure et leur mécanisme d’activation, leur pharmacologie et leur signalisation, jusqu’au développement de nouveaux ligands innovants (mini-anticorps de camélidés et composés photo-activables) et à leurs applications dans plusieurs modèles de maladies du cerveau.

 IGF Nord 223
IGF Nord 223 04 34 35 92 78
04 34 35 92 78-
2009HDR - Biochimie - Université de Montpellier - France
-
1998Doctorat - Biochimie - Université Paris 5 - France
-
1993Master Biochimie - Université Paris 5 - France
-
2009HDR - Biochimie - Université de Montpellier - France
-
1998Doctorat - Biochimie - Université Paris 5 - France
-
1993Master Biochimie - Université Paris 5 - France
-
2011-Directeur de Recherche INSERM - Institut de Génomique Fonctionnelle - Montpellier - France
-
2001-2010Chargé de Recherche INSERM - Institut de Génomique Fonctionnelle - Montpellier - France
-
1998-2001Post-doctorat (Dir. H. Bourne) - Université de Californie - San Francisco - USA
-
1993-1998Thèse (Dir. H. Bedouelle) - Institut Pasteur - Paris - France
- Distinctions
-
2024Prix de l'innovation « Coup de cœur » - Université de Montpellier
-
2023Prix de l'Académie Nationale de Médecine (prix ANM), Paris
-
2022Prix de l'Académie des Sciences (Prix Velluz), Paris
-
2021Prix du chercheur du fleuve Yangtze, ministère de l'éducation, Chine
-
2013-Professeur invité, Université des sciences et technologies de Huazhong, Wuhan, Chine
-
2013"Bell Prize" pour les experts étrangers dans la province de Hubei, Chine
-
2001Bourse post-doctorale - Fondation pour la recherche médicale (FRM)
-
1999Bourse post-doctorale - Bourse à long terme Human Frontier Science Program (HFSP).
-
1998Bourse post-doctorale - Association pour la recherche sur le cancer (ARC)
-
1993Bourse pré-doctorale du Ministère français de la Recherche
- Responsabilités
-
2021-Chef d'équipe - Institut de génomique fonctionnelle - France
-
2008-Directeur scientifique, équipe collaborative - CNRS-Cisbio Revvity
-
2008-Co-directeur du laboratoire sino-français de signalisation cellulaire, College of Life Science, Huazhong University of Science and Technology - Wuhan - Chine

 IGF Nord 209b
IGF Nord 209b 04 34 35 92 78
04 34 35 92 78-
2016Doctorat en Biochimie - Université Pierre et Marie Curie Paris 6, Institut Pasteur - Paris - France
-
2012Master en Biochimie & Biologie Moléculaire - Université Pierre et Marie Curie Paris 6 - France
-
2010Licence en Biochimie & Biologie Moléculaire - Université Denis Diderot Paris 7 - France
-
2023Chargée de recherche CNRS - Institut de Génomique Fonctionnelle - Montpellier - France
-
2021-2023Postdoctorante - Institut de Génomique Fonctionnelle - Montpellier - France
-
2017-2021Postdoctorante - Imperial College London - UK
-
2016-2017Postdoctorante - Institut Pasteur - Paris - France
-
2012-2016Doctorante - Institut Pasteur - Paris - France
-
2011Stage de 6 mois - University College London and Birkbeck - UK
-
2010Stage de 6 mois - Université Denis Diderot Paris 7 - Paris - France
- Distinctions
-
2021-2023Bourse "Retour en France" - Fondation pour la Recherche Médicale (FRM)
-
2012-2015Bourse de thèse - Ministère de la Recherche
- Responsabilités
-
2023Membre du comité organisateur du GDR CNRS sur les protéines membranaires "Appicom"
-
2022-Membre du comité du réseau i-GPCRnet
-
2021-Mentor dans le module doctoral "Femmes et Sciences" - Université de Montpellier - France
- Je suis biochimiste et biophysicienne de formation, spécialisée dans l'étude des protéines membranaires et de leurs interactions avec les lipides.
- Mes recherches portent sur les récepteurs couplés aux protéines G et sur la compréhension des mécanismes moléculaires par lesquels ils fonctionnent et intègrent différents types d'informations de leur environnement. Pour cela, j'utilise principalement la microscopie cryo-électronique en particules uniques et différentes méthodes photo-physiques telles que le FRET résolu en temps.

 IGF Nord 223
IGF Nord 223 04 34 35 92 78
04 34 35 92 78-
2002HDR - Biochimie - Université de Montpellier - France
-
1987Doctorat - Biochimie - Université de Montpellier - France
-
1984Master 2 Biologie Moléculaire - Université de Montpellier - France
-
1983Master 1 Physiologie & Master 1 Biochimie - Univ Pierre et Marie Curie - Paris
-
1981École Normale Supérieure Ulm - Paris
-
2014-Directeur de Recherche CE CNRS - Institut de Génomique Fonctionnelle - Montpellier - France
-
2010-2020Directeur - Institut de Génomique Fonctionnelle - Montpellier - France
-
2003-2015Directeur Dépt Pharmacologie Moléculaire - Institut de Génomique Fonctionnelle - Montpellier - France
-
1996-2014Directeur de Recherche CNRS - CCIPE - Montpellier - France
-
1990-1992Post-doctorat (Dir. S Heinemann) - Salk Institute - San Diego - USA
-
1989-1996Chargé de Recherche CNRS - CCIPE - Montpellier - France
-
1987-1988Service National
-
1985-1987Normalien Doctorant - Univ Montpellier - France
-
1984-1987Doctorat en biologie Moléculaire - Univ Montpellier - France
- Distinctions
-
2022Grand Prix Lamonica de Neurologie, Académie des Sciences
-
2018-2019Highly Cited Researcher, Clarivate, Web of Science Group
-
2015Prix Galien France pour la recherche thérapeutique
-
2014Fait partie des 3000 chercheurs les plus cités au monde - ISI Thomson
-
2013Prix Camille Woringer de la Fondation Recherche Médicale
-
2011Médaille d'Argent du CNRS
-
2011Prix "Coup d'élan" pour la Recherche française Fondation Bettencourt Schuller
-
2010"Bell Prize" for foreigner expert in Hubei province, Chine
-
2009Grand Prix Léon Velluz de l'Académie des Sciences
- Responsabilités
-
2020-2026Vice-Président, puis Président du Conseil Scientifique de la FRC
-
2020-2026Président du SAB de l'IBPS - Paris
-
2010-Directeur de l’équipe collaborative CNRS-Cisbio Revvity
-
2008-2026Co-directeur du laboratoire franco-chinois de Signalisation Cellulaire, College of Life Science, Huazhong University of Science and Technology - Wuhan - Chine
-
2008-2012Membre de la commission scientifique 1 de l'Inserm
-
2006Membre du conseil Scientifique se l'ANR Neuroscience
-
2003-2007Membre du Conseil Scientifique de la FRC
-
2002-2015Membre du Comité de Nomenclature de IUPHAR
-
2000-2004Membre du Comité du Science Program de HFSP
- Mon domaine d’expertise est la neuropharmacologie moléculaire, en particulier l’étude des récepteurs couplés aux protéines G et leur modulation par des ligands innovants.
- Mes intérêts de recherche incluent :
- les récepteurs métabotropes du glutamate et du gaba
- les anticorps simples chaînes de camélidés (nanocorps ou VHH)
- les technologies de transfert d’énergie de résonance (FRET, HTRF, BRET)

 IGF Nord 221a
IGF Nord 221a 04 34 35 93 07
04 34 35 93 07

 IGF Nord 219
IGF Nord 219 04 34 35 93 13
04 34 35 93 13

 IGF Nord 221a
IGF Nord 221a 04 34 35 93 07
04 34 35 93 07-
2007Diplôme de l’École Pratique des Hautes Études - Spécialité Pharmacologie Cellulaire et Moléculaire - Paris - France
-
2001Certificat d’Études Spécialisées Technologiques - Recherche Biomédicale, option Immunologie - ESTBA - Paris France
-
2000Brevet de technicien supérieur (BTS) - Biochimiste - ESTBA - Paris - France
-
2023-Ingénieure d’Étude CNRS - Institut de Génomique Fonctionnelle - Montpellier - France
-
2018-2022Ingénieure d’Étude CNRS - Institut de Neurosciences Paris-Saclay - Gif sur Yvette - France
-
2014-2018Assistante Ingénieure CNRS - Institut de Neurosciences Paris-Saclay - Gif sur Yvette - France
-
2008-2014Assistante Ingénieure CNRS - UMR 8147 - Paris - France
-
2022-2008Technicienne de Recherche CNRS - UMR 8147 - Paris - France
-
2002Technicienne de Recherche CDD INSERM - U25 - Paris - France
-
2023-Co-responsable du laboratoire L2 Nord - Institut de Génomique Fonctionnelle - Montpellier
- Je suis ingénieure d’étude au CNRS. Mes centres d’intérêt couvrent :
- Neurobiologie
- Immunologie
- Techniques de laboratoire
- Cultures de lignées cellulaires et cultures primaires
- Biologie moléculaire : PCR, Extraction, tests biochimiques
- Production de nanobodies
- Travail en secteur L2
- Marquages et tri cellulaire
- Dosages protéiques
- Analyse de données
- GraphPad Prism
- Maîtrise du Pack office
- Elphy, Labscribe, Deep Lab Cut

 IGF Nord 223
IGF Nord 223 04 34 35 93 07
04 34 35 93 07
-
2022Master - Biologie Santé - Neurosciences - Université de Montpellier - France
-
2020Licence Physiologie Animale et Neurosciences - Université Montpellier - Montpellier - France
-
2022-Doctorant (Dir. P. Rondard & V. Perrier) – Institut de Génomique Fonctionnelle (IGF) & Institut des Neurosciences de Montpellier (INM) - Montpellier - France
-
2022Stage de recherche de Master 2 (Dir. V. Perrier) - Institut des Neurosciences de Montpellier (INM) - Montpellier – France
-
2021-2022Chef de salle / Coordinateur – Centre de Vaccination de l’Hôtel de Ville de Montpellier - Montpellier - France
-
2021Stage de recherche de Master 1 (Dir. K. Loulier) - Institut des Neurosciences de Montpellier (INM) - Montpellier - France
-
2022Parrainage par l’Association France Alzheimer (3 ans)
-
2022Bourse doctorale de l’École Doctorale ED168 - Sciences Chimiques et Biologiques pour la Santé (CBS2)
- Je suis un doctorant spécialisé dans le développement de « Nanobodies » ciblant spécifiquement les récepteurs métabotropes du glutamate (mGlu), en tant que biosenseurs, modulateurs allostériques et outils thérapeutiques innovants dans de multiples neuropathologies. Mon expertise et mes intérêts de recherche se portent vers la pharmacologie sur les récepteurs mGlu, les technologies FRET/HTRF®, ainsi que les maladies neurodégénératives.
- Principales techniques maitrisées
- Bactériologie : transformation, production de nanobody en E. coli
- Cultures de lignées cellulaires (HEK293 ATCC) et transfection (electroporation/lipofection)
- Pharmacologie : technologies FRET/HTRF®, mesures d’affinité et d’activité de ligands
- Biologie moléculaire : qPCR, Western Blot, Extraction d’ADN
- Formation Concepteur de Niveau 1
- Analyse de données
- GraphPad Prism
- Maîtrise du Pack office
- Image J/FIJI

 IGF Nord 221a
IGF Nord 221a 04 34 35 93 07
04 34 35 93 07

 IGF Nord 223
IGF Nord 223 04 34 35 92 78
04 34 35 92 78

 IGF Nord 209b
IGF Nord 209b 04 34 35 93 07
04 34 35 93 07-
2022Doctorat Chimie Biologie - Université Grenoble Alpes - Grenoble - France
-
2018Master Biochimie et Biologie Moléculaire - Université Claude Bernard - Lyon - France
-
2016Licence Biochimie - Université Claude Bernard - Lyon - France
-
2022-Postdoctorante - Institut de Génomique Fonctionnelle - Montpellier - France
-
2018-2022Doctorante - Laboratoires SyMMES & LPCV - Institut de Recherche Interdisciplinaire de Grenoble - Grenoble - France
-
2022-Membre de l’association SP’IN de l’Institut de Génomique Fonctionnelle de Montpellier
-
2019-2021Représentante élue des doctorants de l’École Doctorale Chimie et Sciences du Vivant de Grenoble
-
2018-2020Représentante élue du personnel non permanent au conseil de laboratoire du SyMMES
- Au sein de l’IGF, je m’intéresse aux interactions entre des récepteurs couplés aux protéines G et leur environnement lipidique. Grâce à ma thèse au SyMMES et à mon post doc à l’IGF, j’ai des compétences en production et purification de protéines solubles et membranaires dans des systèmes procaryotes et eucaryotes. Par ailleurs, l’étude des interactions entre les protéines et/ou avec leur environnement se fait notamment grâce à des techniques de microscopie électronique et de FRET.

 IGF Nord 209b
IGF Nord 209b 04 34 35 92 89
04 34 35 92 89-
2019Doctorat Neurosciences - Université Montpellier - Montpellier - France
-
2014Master Biologie Santé - Endocrinologie et Neurosciences - Université Montpellier - Montpellier - France
-
2012Licence Physiologie Animale et Neurosciences - Université Montpellier - Montpellier - France
-
2022-Chercheur CDD (Dir. Dr P. Rondard) - Institut de Génomique Fonctionnelle (IGF) - Montpellier - France
-
2019-2021Chercheur CDD (Dir. Pr J.F. Liu) - Université des Sciences et des Technologies de Huazhong - Wuhan - Chine
-
2015-2019Doctorant (Dir. Dr V. Perrier) - Laboratoire Mécanismes Moléculaires dans les Démences Neurodégénératives (MMDN) - Montpellier - France
-
2014Stage de 6 mois (Dir. Dr V. Perrier) - Laboratoire Mécanismes Moléculaires dans les Démences Neurodégénératives (MMDN) - Montpellier - France
-
2013Stage de 2 mois (Dir. Dr A. Pattyn)- Institut des Neurosciences de Montpellier (INM) - Montpellier - France
-
2012Stage de 2 mois (Dr. Y. Bessin) - Centre de Biologie Structurale (CBS) - Montpellier - France
- Distinctions
-
2022-2024Bourse de postdoctorat du labex MAbImprove
-
2020Prix de thèse de l'Académie Nationale de Pharmacie
-
2018Bourse de 4ème année de thèse - Centre d'excellence en Maladies Neurodégénératives (CoEN)
-
2015-2018Bourse de thèse - Ministère de la Recherche
- Responsabilités
-
2024-Membre élu du conseil de laboratoire - Institut de Génomique Fonctionnelle
-
2021-Membre du comité du réseau i-GPCRnet
- Je suis un chercheur postdoctoral dont les projets dans lesquels je suis associé visent à développer des nanobodies ciblant les récepteurs mGlu comme immunothérapie innovante pour le traitement de maladies du cerveau. Grâce à mes différentes expériences, mon expertise couvre les domaines de la neuropharmacologie, les technologies de transfert d’énergie de résonance de type Förster (FRET, HTRF®), les récepteurs métabotropes du glutamate (mGlu) et les maladies neurodégénératives.

 IGF Nord 223
IGF Nord 223 04 34 35 92 89
04 34 35 92 89

 IGF Nord 215a
IGF Nord 215a 04 34 35 92 78
04 34 35 92 78

 IGF Nord 215b
IGF Nord 215b 04 34 35 92 78
04 34 35 92 78-
2023Ingénieur en biotechnologies – Biotechnologies – Sup’Biotech – France
-
2024-Thèse Cifre en pharmacologie moléculaire et biochimie (Dir. J-P. Pin / P. Rondard) – Institut de Génomique Fonctionnelle, CNRS UMR 5203, Neurorécepteurs, dynamique et fonctions / Revvity life sciences - Montpellier - France
-
2023-2024Ingénieur d’études (CDD) - Institut de Génomique Fonctionnelle, CNRS UMR 5203, Neurorécepteurs, dynamique et fonctions - Montpellier - France
-
2023Stage ingénieur (6 mois) - Institut de Génomique Fonctionnelle, CNRS UMR 5203, Neurorécepteurs, dynamique et fonctions - Montpellier – France
-
2021Stage assistant ingénieur (4 mois) - Institut de Génomique Fonctionnelle, UMR 5203, Neuroprotéomique et signalisation des pathologies cérébrales / Neurobiobomics - Montpellier - Fance
-
2021Stage technicien de laboratoire (2 mois) - Université Paris-cité, UMR-S 1139, physiopathologie et pharmacotoxicologie placentaire humaine : Microbiote pré & post natal - Paris - France
-
2024-Bourse de these Cifre - ANRT
- Je suis doctorant en thèse Cifre travaillant avec Revvity et l’IGF dans le laboratoire eidos, développant des essais HTRF innovants pour une meilleure compréhension de la signalisation des GPCR.
- Mes recherches impliquent :
- Développement d’essais basés sur la technologie HTRF
- Pharmacologie : Caractérisation de ligands de GPCR, Mesure d’affinité d’IgG, Signalisation des protéines G
- Biochimie : immunomarquage, purification de protéine
- Criblage à haut débit
- Culture cellulaire: culture de lignées stables, transfection, préparation de membranes

 IGF Nord 215b
IGF Nord 215b 04 34 35 92 78
04 34 35 92 78

 IGF Nord 223
IGF Nord 223 04 34 35 93 07
04 34 35 93 07-
2023Master, chimie moléculaire - Université de Rennes 1 - Rennes - France.
-
2020-2023École Nationale Supérieur de Chimie de Rennes (ENSCR), biotechnologie - Université de Rennes 1 - Rennes - France
-
2017-2020Classe Préparatoire aux Grandes Écoles, physique-chimie - Toulon - France
-
2024-Doctorant (Dir. P. Rondard & L. Prézeau) - Institut de Génomique Fonctionnelle (IGF) - Montpellier - France
-
2022-2023Ingénieur d'étude (Dir. P. Rondard) - Institut de Génomique Fonctionnelle (IGF) - Montpellier - France
-
2023Stage de recherche Master 2 (Dir. A. Kapus) - Unity Health Toronto (UHT) - Toronto - Canada
-
2022Stage de recherche Master 1 (Dir. M. Suskiewicz) - Centre de Biophysique Moléculaire (CBM) - Orléans - France
-
2024Bourse doctorale de l’École Doctorale ED168 - Sciences Chimiques et Biologiques pour la Santé (CBS2)
- Je suis un doctorant spécialisé dans le développement de « Nanocorps » (VHH) ciblant les récepteurs couplés aux protéines G (RCPG) et plus précisément les récepteurs métabotropes du glutamate (mGluR), avec des applications en tant qu’outils thérapeutiques novateurs pour certaines pathologies du système nerveux central. Mes recherches et mon expertise se concentrent sur de la pharmacologie de ces récepteurs mGluR
- Technique de laboratoire :
- Pharmacologie : transfection de lignées cellulaires (électroporation/lipofection), technologie FRET/HTRF, mesure d’affinité et d’activité de ligand ; immunofluorescence
- Biochimie des protéines : production bactérienne (E. coli) et eucaryote (HEK293), chromatographie d’affinité, gel filtration (FPLC ÄKTA), marquage, spectrométrie de masse, cristallographie
- Biologie moléculaire : PCR, extraction d’ADN
- Chimie organique : chimie click, synthèse chimique de protéines, chimie radicalaire, HPLC, CPG, RMN 1D et 2D
- Formation conception et réalisation de projets et procédures expérimentales (rongeurs et lagomorphes)
- Analyse de données :
- GraphPad Prism
- Pack Office
- Image J

 IGF Nord 223
IGF Nord 223 04 34 35 92 78
04 34 35 92 78

 IGF Nord 216a
IGF Nord 216a 04 34 35 92 63
04 34 35 92 63

 IGF Nord 223
IGF Nord 223 04 34 35 92 78
04 34 35 92 78

 IGF Nord 223
IGF Nord 223 04 34 35 92 78
04 34 35 92 78

 IGF Nord 221a
IGF Nord 221a 04 34 35 92 89
04 34 35 92 89

 IGF Nord 221a
IGF Nord 221a 04 34 35 93 07
04 34 35 93 07
-
2025Master - Biologie Santé, Neurosciences - Université de Montpellier - France
-
2023Licence 3 - Physiologie Animale et Neuroscience - Université de Montpellier - France
-
2022DUT - Génie Biologique - IUT de Montpellier - France
-
2025-Ingénieur CDD CNRS - Institut de Génomique Fonctionnelle - Montpellier - France
-
2025Stagiaire (6 mois) - Institut de Génomique Fonctionnelle - Montpellier - France
-
2024Stagiaire (5 mois) - Institut de Génomique Fonctionnelle - Montpellier - France
-
2022Stagiaire (2 mois et demi) - Institut des Neurosciences de Montpellier - Montpellier - France

 IGF Nord 209b
IGF Nord 209b 04 34 35 92 89
04 34 35 92 89

 IGF Nord 209b
IGF Nord 209b 04 34 35 92 89
04 34 35 92 89-
2020Master - Sciences du Médicament, parcours Biologie Structurale et Conception de Molécules Bioactives - Université de Montpellier - France
-
2018Licence - Biochimie - Université Claude Bernard - Lyon - France
-
2020-Ingénieur CNRS (CDD) - Institut de Génomique Fonctionnelle - Montpellier - France
- Je suis une ingénieure de recherche en biologie cellulaire spécialisée en essais par fluorescence (HTRF) dans le but d’étudier les RCPGs et protéines G.
- Techniques de laboratoire
- Bactériologie
- Cultures de lignées cellulaires
- Marquage d’anticorps
- Biologie moléculaire : qPCR, Western Blot
- Travail en secteur BSL2
- Analyse de données
- GraphPad Prism
- Maîtrise du Pack Office

 IGF Nord 209b
IGF Nord 209b 04 34 35 92 78
04 34 35 92 78

 IGF Nord 209b
IGF Nord 209b 04 34 35 92 78
04 34 35 92 78
- Xu C, Zhou Y, Liu Y, Lin L, Liu P, Wang X, Xu Z, Pin JP*, Rondard P*, Liu J* (2024). Specific pharmacological and Gi/o protein coupling properties of native GPCRs in neurons. Nat Comm 2024 15(1) :1990. doi: 10.1038/s41467-024-46177-z. PMID: 38443355 * Co-corresponding author.
- Lecat-Guillet N, Quast RB, Liu H, Moeller TC, Rovira X, Soldevila S, Lamarque L, Trinquet E, Liu J, Pin JP, Rondard P*, Margeat E*. Concerted conformational changes control metabotropic glutamate receptor activity. Sci Adv 2023 9 (22) :eadf1378. doi: 10.1126/sciadv.adf1378. PMID: 37267369 * Co-corresponding author.
- Meng J, Xu C, Lafon PA, Roux S, Mathieu M, Zhou R, Scholler P, Blanc E, Becker JAJ, Le Merrer J, González-Maeso J, Chames P, Liu J*, Pin JP*, Rondard P*. Optical biosensors of native membrane protein complexes reveal a high proportion of mGlu heterodimers in the brain. Nat Chem Biol 2022, 18, 894-903. doi: 10.1038/s41589-022-01050-2. PMID: 35681029 * Co-corresponding author.
- Liu J, Tang H, Xu C, Zhou S, Zhu X, Li Y, Prézeau L, Xu T, Pin JP*, Rondard P*, Ji W*, Liu J* (2022) Biased signaling due to oligomerization of a G protein-coupled receptor. Nat Comm 2022 13, 6365. doi: 10.1038/s41467-022-34056-4. PMID: 36289206 * Co-corresponding author.
- Haubrich J, Font J, Goupil-Lamy A, Scholler P, Nevoltris D, Acher F, Chames P, Rondard P, Prézeau L*, Pin J-P*. A nanobody activating metabotropic glutamate receptor 4 discriminates between homo- and heterodimers. Proc Natl Acad Sci U S A 2021 118 (33):e2105848118. doi: 10.1073/pnas.2105848118. PMID: 34385321* Co-corresponding author.
- Cao AM, Quast RB, Fatemi F, Rondard P, Pin JP*, Margeat E* (2021) Allosteric modulators enhance agonist efficacy by increasing the residence time of a GPCR in the active state. Nat Comm 2021 12 (1):5426. doi: 10.1038/s41467-021-25620-5. PMID: 34521824* Co-corresponding author.
- Liu H, Yi P, Zhao W, Wu Y, Acher F, Pin JP*, Liu J*, Rondard P. Illuminating the allosteric modulation of the calcium sensing receptor. Proc Natl Acad Sci U S A 2020 117 (35) 21711-21722. doi: 10.1073/pnas.1922231117. PMID: 32817431 * Co-corresponding author.
- Xue L, Sun Q, Zhao H, Rovira X, Gai S, He Q, Pin JP*, Liu J*, Rondard P. Rearrangement of the transmembrane domain interfaces associated with the activation of a GPCR hetero-oligomer. Nat Commun 2019 10, 2765. doi: 10.1038/s41467-019-10834-5. PMID: 31235691 * Co-corresponding author.
- Scholler P, Nevoltris D, De Blundel D, Bossi S, Moreno D, Rovira X, Møller TC, El-Moustaine D, Mathieu M, Blanc E, McLean H, Dupuis E, Mathis G, Trinquet E, Daniel H, Valjent E, Baty D, Chames P, Rondard P*, Pin JP*. Allosteric nanobodies uncover a role of hippocampal mGlu2 receptor homodimers in contextual fear consolidation. Nat Commun 2017 8 (1):1967. doi: 10.1038/s41467-017-01489-1. PMID: 29213077 * Co-corresponding author.
- Scholler P, Moreno D, Lecat-Guillet N, Doumazane E, Monnier C, Charrier-Savournin F, Fabre L, Chouvet C, Soldevila S, Lamarque L, Donsimoni G, Roux T, Zwier JM, Trinquet E, Rondard P*, Pin JP*. HTS-compatible FRET-based conformational sensors clarify membrane receptor activation. Nat Chem Biol 2017 13, 372-380. doi: 10.1038/nchembio.2286. PMID: 28135236 * Co-corresponding author
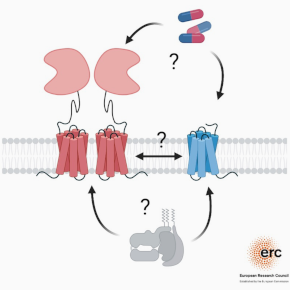
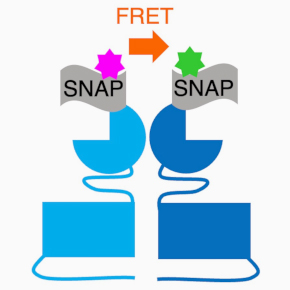
Développement de méthodes et d’outils pour étudier les RCPG
Responsable
Jean-Philippe PIN
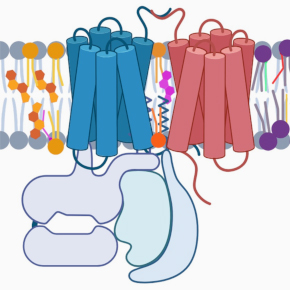
Complexes de récepteurs mGlu et leur dynamique en environnement natif
Responsable
Laurent PREZEAU

Nanobodies ciblant les récepteurs mGlu : immunothérapie innovante pour le traitement des maladies du cerveau
Responsable
Philippe RONDARD

Revvity Cisbio
Codolet, France

Emmanuel Margeat
Centre de Biologie Structurale (CBS), Montpellier, France

Francine Acher
Université Paris Cité, Paris, France

Patrick Chames
Aix-Marseille Université, France

Jianfeng Liu
Huazhong University of Science and Technology (HUST), Wuhan, China
Amadeu Llebaria
University of Barcelona, Barcelona, Spain

Doctorants
• Alexandre Bouyssou (Genentech, USA)
• Laetitia Comps-Agrar (Genentech, USA)
• Mélanie Da Silva
• Damien Maurel
• Carine Monnier
• Mathieu Oosterlaken
• Marie-Laure Rives
• Pauline Scholler (Revvity)
Post-doctorants
• Fanny Dubois
• Driss El Moustaine
• Nathalie Lecat-Guillet (Revvity)
• Oualid Sbai
• Li Xue (Shanghai Jiao Tong University School of Medicine, Shanghai, China)
Ingénieurs
• Emilie Blanc
• Michaël Mathieu (Revvity)
• Jessica Monnic (Mabqi)
• Roxane Perrin
• Salomé Roux




