Équipe Laurent JOURNOT
Génomique fonctionnelle des gènes soumis à empreinte parentale

Notre équipe est un laboratoire multidisciplinaire qui combine la biologie moléculaire, la biologie des cellules souches, la génomique et les statistiques pour étudier les fonctions des gènes soumis à empreinte parentale. Nous nous concentrons sur 1- la fonction du facteur de transcription ZAC1/PLAGL1, 2- le rôle et la régulation du réseau des gènes soumis à empreinte, et 3- l’impact de ces gènes sur le développement cortical. A travers l’étude des gènes soumis à empreinte, nous développons des méthodes statistiques pour analyser les biais alléliques dans les données transcriptomiques.
L’empreinte génomique parentale est un mécanisme épigénétique de régulation des gènes qui limite l’expression d’un gène à un allèle en fonction de son origine parentale. L’empreinte génomique est essentielle au développement des mammifères. Elle cible environ 150 gènes chez les mammifères placentaires (euthériens) et moins d’une douzaine chez les marsupiaux (métathériens) ; elle n’affecte pas les mammifères ovipares (protothériens) et les autres clades (oiseaux, reptiles, amphibiens, poissons…).
Les mécanismes qui expliquent l’expression mono-allélique dépendant de l’origine parentale des gènes soumis à empreinte ont été étudiés en détail (Hanna & Kelsey, Heredity (Edinb), 2014 ; Ferguson-Smith, Nat Rev Genet, 2011). Un grand nombre de gènes soumis à empreinte sont regroupés dans un nombre limité de loci. La répression de l’un de leurs allèles parentaux est contrôlée par les régions de contrôle de l’empreinte (ICR), qui comprennent généralement une région différentiellement méthylée (DMR). La (dé)méthylation des DMR paternelles et maternelles régit l’expression ou la répression des gènes soums à empreinte voisins.
Les défauts d’empreinte à des loci définis entraînent différents syndromes aux phénotypes complexes : Silver-Russel, Angelman, Prader-Willi, Beckwith-Wiedemann et TNDM (Transient Neonatal Diabetes Mellitus). En outre, un certain nombre de gènes soumis à empreinte sont impliqués dans la formation de tumeurs en tant qu’oncogènes ou gènes suppresseurs de tumeurs.
Nous avons montré que les gènes soumis à empreinte appartiennent à un réseau de gènes corégulés que nous avons appelé le réseau des gènes soumis à empreinte (IGN) (Varrault et al., Dev. Cell, 2006). Nous avons ensuite découvert que l’IGN est régulé lors de la transition de la prolifération à la quiescence et à la différenciation pendant le retrait du cycle cellulaire des fibroblastes, l’adipogenèse in vitro et la régénération musculaire in vivo. L’IGN comprend également des gènes exprimés de façon bi-allélique, notamment des gènes contrôlant la composition de la matrice extracellulaire. Nos observations suggèrent que les gènes imprintés sont impliqués dans un processus biologique commun qui pourrait expliquer leurs rôles apparemment divers dans le développement embryonnaire, l’obésité, le diabète, la physiologie musculaire et les néoplasmes (Al Adhami et al., Genome Res., 2015).

Progéniteurs neuraux positifs à la nestine (vert) générés à partir de cellules souches embryonnaires uniparentales de souris exprimant le marqueur cortical Pax6 (rouge) et colorés au DAPI (bleu).

 IGF Sud 022
IGF Sud 022 04 34 35 92 40
04 34 35 92 40-
1991Doctorat - Biologie moléculaire - Université de Montpellier - France
-
1986Master - Biologie Santé - Université de Montpellier - France
-
1985Agrégation de Biochimie-Physiologie
-
1998-Directeur de Recherche CNRS - Institut de Génomique Fonctionnelle - Montpellier - France
-
1992-1998Chargé de Recherche CNRS - Institut de Génomique Fonctionnelle - Montpellier - France
-
1991-1992Postdoctorat - Université d’Heidelberg - Heidelberg - Allemagne
-
1987-1991Thèse - Centre CNRS INSERM de Pharmacologie-Endocrinologie - Montpellier - France
- Prix/Distinctions
-
2000Prix de la Fondation Schlumberger pour l’Éducation et la Recherche
-
1997Prix de la Ligue Nationale contre le Cancer
-
1995Médaille de Bronze du CNRS
-
1991Bourse de l’EMBO
- Responsabilités scientifiques
-
2011-2020Directeur de l’UMS BioCampus Montpellier
-
2000-2024Responsable de la plateforme MGX-Montpellier GenomiX
-
2007-2026Responsable de l’équipe Génomique Fonctionnelle des Gènes Soumis à Empreinte Parentale - IGF Montpellier
-
1998-2007Responsable de l’équipe Prolifération-Différenciation-Apotpose - IGF Montpellier
- Je suis biologiste moléculaire, spécialisé dans la génération de modèles de pathologies humaines et dans leur analyse par des méthodes informatiques et statistiques.
- Mes intérêts de recherche incluent :
- L’empreinte génomique parentale
- Le contrôle génétique de la prolifération, la quiescence et la différenciation.
- L’épigénétique, la transcriptomique, les statistiques appliquées aux données de génomique

 IGF Sud 022
IGF Sud 022 04 34 35 92 40
04 34 35 92 40-
2023HDR - Université de Montpellier - France
-
2002Doctorat Biologie Santé - Université de Montpellier - France
-
2009-Chargé de Recherche INSERM - Institut de Génomique Fonctionnelle - Montpellier - France
-
2006-2009Postdoctorat - Université libre de Bruxelles - Belgique
-
2002-2006Postdoctorat - Université de Bristol - Royaume uni
-
1998-2022Thèse - Centre CNRS INSERM de Pharmacologie-Endocrinologie - Montpellier - France
-
2008-2009Bourse Marie Sklodowska-Curie
- Les gènes soumis à empreinte sont des gènes au profil d’expression original puisqu’un seul des deux allèles parentaux est transcrit. Ces gènes, dont une seule copie est active, sont impliqués dans de nombreux troubles du développement dont l’étiologie demeure peu connue.
- Pour mieux cerner le rôle des gènes soumis à empreinte pendant le développement neurologique, nous modifions le génome en utilisant des outils basés sur CRISPR/Cas9 (CRISPR activation et inhibition, CRISPR édition pour générer des protéines tronquées...) sur des cerveaux générés in vitro à partir de cellules souches embryonnaires de souris.
- Nous espérons améliorer notre compréhension de la fonction des gènes soumis à empreinte au cours du développement du cerveau.
- Mes intérêts de recherche incluent :
- Génétique du développement
- Bio-ingénierie
- Maladies rares

 Fac de Pharma
Fac de Pharma 04 11 75 96 83
04 11 75 96 83

 Fac de Pharma
Fac de Pharma


 IGF Sud 022
IGF Sud 022 04 34 35 92 40
04 34 35 92 40

 Fac de Pharma
Fac de Pharma 04 11 75 96 82
04 11 75 96 82

 IGF Sud 022
IGF Sud 022 04 34 35 92 40
04 34 35 92 40-
2015Ingénieur en Génie Biologique - EICNAM - Montpellier - France
-
2009Licence en Génie Biologique - CNAM - Montpellier - France
-
2002Année de spécialisation en Recherche Biomédicale - ESTBA - Paris
-
2001DUT en Analyses Biologiques et Biochimiques (ABB) - IUT - Montpellier - France
-
1999Baccalauréat S-SVT - Lycée Jeanne d’Arc - Millau - France
-
2024-Ingénieur (IE) - Institut de Génomique Fonctionnelle (IGF) - CNRS Montpellier - France
-
2015-2024Assistante-Ingénieur (AI) - IGF - CNRS Montpellier - France
-
2012-2015Technicienne de classe exceptionnelle (TCE) - IGF - CNRS Montpellier - France
-
2002-2011Technicienne (TCN) fonctionnaire - IGF - CNRS Montpellier - France
-
2001Technicienne de laboratoire (CDD 2 mois) - Centre Hospitalier du Sud-Aveyron - Saint-Affrique - France
-
2025-Assistant de prévention de l'IGF
-
2020-2025Membre de la commission équipement de l’IGF
-
2010-Responsable d’appareils communs (dont lecteurs de plaques) à l’IGF
-
2007-Responsable du parc des appareils de PCR quantitative de l’IGF
- Je suis ingénieur dans une équipe de recherche. Mon travail s’organise autour de deux axes :
- Partie scientifique : conduire un ensemble de techniques spécialisées en biologie cellulaire, moléculaire et en génomique dans le cadre des projets de l’équipe
- Partie management : formations dans l’équipe et pour les appareils communs dont je suis responsable, tutorats d’étudiants en BTS, gestion des stocks et des commandes, gestion de bases de données (sous excel), …
- Techniques de laboratoire
- Cultures de lignées cellulaires et de cellules primaires, humaines et murines
- Biologie moléculaire : PCR, PCR quantitative, Clonage (sous-clonage, Gibson Assembly, CRISPR/Cas9, …), Western Blot, …
- Biologie cellulaire : Immunofluorescence, Cytométrie en flux, Microscopie, …
- Analyses de données
- Logiciels spécifiques à mon activité : ZEN, ApE, SnapGene, Flowing Software, Geslab, PrimerBLAST,
- GraphPad - Prism
- Image J

 IGF Sud 022
IGF Sud 022 04 34 35 92 40
04 34 35 92 40

 IGF Sud 022
IGF Sud 022 04 34 35 92 40
04 34 35 92 40
- Kumar P, Courtes M, Lemmers C, Le Digarcher A, Coku I, Monteil A, Hong C, Varrault A, Liu R, Wang L, Bouschet T. Functional mapping of microRNA promoters with dCas9 fused to transcriptional regulators. Front Genet. 2023 May5;14:1147222. doi: 10.3389/fgene.2023.1147222. PMID: 37214422
- Janbain A, Reynès C, Assaghir Z, Zeineddine H, Sabatier R, Journot L. TopoFun: a machine learning method to improve the functional similarity of gene co-expression modules. NAR Genom Bioinform. 2021 Nov 8;3(4):lqab103. doi: 10.1093/nargab/lqab103. PMID: 34761220
- Reynès C, Kister G, Rohmer M, Bouschet T, Varrault A, Dubois E, Rialle S, Journot L, Sabatier R. ISoLDE: a data-driven statistical method for the inference of allelic imbalance in datasets with reciprocal crosses. 2020 Jan 15;36(2):504-513. doi: 10.1093/bioinformatics/btz564. PMID: 31350542
- Varrault A, Journot L, Bouschet T. Cerebral Cortex Generated from Pluripotent Stem Cells to Model Corticogenesis and Rebuild Cortical Circuits: In Vitro Veritas? Stem Cells Dev. 2019 Mar 15;28(6):361-369. doi: 10.1089/scd.2018.0233. PMID: 30661489.
- Baudement MO, Cournac A, Court F, Seveno M, Parrinello H, Reynes C, Sabatier R, Bouschet T, Yi Z, Sallis S, Tancelin M, Rebouissou C, Cathala G, Lesne A, Mozziconacci J, Journot L, Forné T. High-salt-recovered sequences are associated with the active chromosomal compartment and with large ribonucleoprotein complexes including nuclear bodies. Genome Res. 2018 Nov;28(11):1733-1746. doi: 10.1101/gr.237073.118. PMID: 30287550
- Varrault A, Eckardt S, Girard B, Le Digarcher A, Sassetti I, Meusnier C, Ripoll C, Badalyan A, Bertaso F, McLaughlin KJ, Journot L, Bouschet T. Mouse Parthenogenetic Embryonic Stem Cells with Biparental-Like Expression of Imprinted Genes Generate Cortical-Like Neurons That Integrate into the Injured Adult Cerebral Cortex. Stem Cells. 2018 Feb;36(2):192-205. doi: 10.1002/stem.2721. PMID: 29044892
- Varrault A, Dantec C, Le Digarcher A, Chotard L, Bilanges B, Parrinello H, Dubois E, Rialle S, Severac D, Bouschet T, Journot L. Identification of Plagl1/Zac1 binding sites and target genes establishes its role in the regulation of extracellular matrix genes and the imprinted gene network. Nucleic Acids Res. 2017 Oct 13;45(18):10466-10480. doi: 10.1093/nar/gkx672. PMID: 28985358
- Bouschet T, Dubois E, Reynès C, Kota SK, Rialle S, Maupetit-Méhouas S, Pezet M, Le Digarcher A, Nidelet S, Demolombe V, Cavelier P, Meusnier C, Maurizy C, Sabatier R, Feil R, Arnaud P, Journot L, Varrault A. In Vitro Corticogenesis from Embryonic Stem Cells Recapitulates the In Vivo Epigenetic Control of Imprinted Gene Expression. Cereb Cortex. 2017 Mar 1;27(3):2418-2433. doi: 10.1093/cercor/bhw102. PMID: 27095822.
- Al Adhami H, Evano B, Le Digarcher A, Gueydan C, Dubois E, Parrinello H, Dantec C, Bouschet T, Varrault A, Journot L. A systems-level approach to parental genomic imprinting: the imprinted gene network includes extracellular matrix genes and regulates cell cycle exit and differentiation. Genome Res. 2015 Mar;25(3):353-67. doi: 10.1101/gr.175919.114. Epub 2015 Jan 22. PMID: 25614607
- Varrault A, Gueydan C, Delalbre A, Bellmann A, Houssami S, Aknin C, Severac D, Chotard L, Kahli M, Le Digarcher A, Pavlidis P, Journot L. Zac1 regulates an imprinted gene network critically involved in the control of embryonic growth. Dev Cell. 2006 Nov;11(5):711-22. doi: 10.1016/j.devcel.2006.09.003. PMID: 17084362.

Caractérisation fonctionnelle du facteur de transcription ZAC1/PLAGL1
Responsables
Annie VARRAULT et Laurent JOURNOT
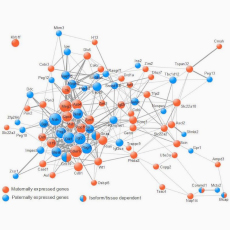
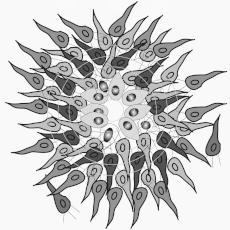
Rôle des gènes soumis à empreinte parentale dans le neurodéveloppement
Responsable
Tristan BOUSCHET
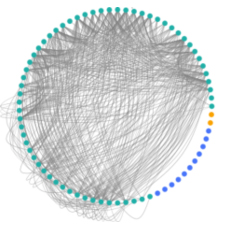
Méthodes statistiques pour les données de génomique
Responsables
Christelle REYNES et Laurent JOURNOT




