Équipe Sébastien GRANIER – Bernard MOUILLAC
Structure et fonction des protéines membranaires pour l’innovation thérapeutique


La signalisation cellulaire est fortement régulée par des protéines exprimées au niveau de la membrane plasmique. Ces protéines membranaires jouent ainsi un rôle clé dans la physiologie humaine normale et pathologique et ont un fort potentiel thérapeutique. Notre recherche vise d’abord à élucider les fondements moléculaires qui régissent la fonction de ces protéines. Ces connaissances sont ensuite utilisées pour développer des molécules pouvant moduler leur activité et leur fonction, un point de départ pour le développement de médicaments innovants.
Dans ce contexte, notre recherche vise à élucider les bases moléculaires de la liaison et de la régulation de l’activité de ces protéines par des approches intégrées de pharmacologie moléculaire et de biologie structurale combinant la biochimie, la RMN, la cristallographie, la cryoEM, la spectrométrie de masse structurale (native et HDX) et la bioinformatique. Nous nous intéressons plus particulièrement à des protéines qui possèdent un fort potentiel pour le développement de molécules à visées thérapeutiques comme les récepteurs couplés aux protéines G (GPCRs) et les enzymes contrôlant l’homéostasie des céramides. Forts de ces connaissances, nous utilisons des approches de chemo-informatique et notamment l’IA, pour identifier des molécules pouvant moduler l’activité et la fonction de ces protéines. Ces molécules constituent à la fois des outils pour étudier la fonction des protéines d’intérêts in vivo et aussi un point de départ pour le développement de molécules à visées thérapeutiques.
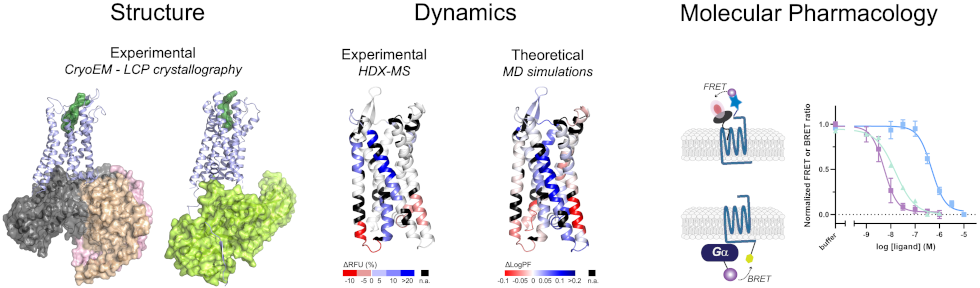
Structure, dynamique et pharmacologie moléculaire de complexes de signalisation de récepteurs couplés aux protéines G (GPCRs) obtenues par cryo-microscopie électronique ou crystallographie en phase cubique lipidique (CryoEM ou LCP), échange Hydrogene/Deuterium couplé à la spectrométrie de masse (HDX-MS), simulations de dynamique moléculaire (MD) et expériences de FRET ou BRET sur cellules vivantes.

 IGF Nord 204a
IGF Nord 204a 04 34 35 92 80
04 34 35 92 80
-
2004Doctorat - Pharmacologie moléculaire - Interface Chimie Biologie - Université de Montpellier - France
-
2001Master - Interface Chimie Biologie - Université de Montpellier - France
-
2015-Directeur de Recherches INSERM - Institut de Génomique Fonctionnelle - Montpellier - France
-
2012-2015Chargé de Recherches INSERM - Institut de Génomique Fonctionnelle - Montpellier - France
-
2010-2012Chargé de Recherches - Stanford University - Stanford - USA
-
2007-2010Chargé de Recherches INSERM - Institut de Génomique Fonctionnelle - Montpellier - France
-
2005-2007Post-doctorat (Dir. B. Kobilka) - Stanford University - Stanford - USA
- Distinctions
-
2019Prix de l'Académie des Sciences Mme Victor Noury née Catherine Langlois - Fondation de l'Institut de France.
-
2015-2020ERC consolidator grant ADIPOR
- Responsabilités
-
2015-Responsable d'équipe - Institut de Génomique Fonctionnelle - Montpellier - France
- Formé à l'interface chimie-biologie, je me suis spécialisé dans l'étude des mécanismes moléculaires qui régissent la fonction des récepteurs couplés aux protéines G par des approches intégrées de biologie structurale et de pharmacologie moléculaire.

 IGF Sud 001
IGF Sud 001 04 34 35 92 68
04 34 35 92 68

 IGF Sud 025
IGF Sud 025 04 34 35 93 24
04 34 35 93 24-
2012Doctorat en Biochimie - Université Pierre et Marie Curie - Paris - France
-
2009Master "Protéomique Structurale et Fonctionnelle" - Université Pierre et Marie Curie - Paris - France
-
2008Maîtrise en Biochimie - Université Libanaise - Beyrouth - Liban
-
2023-Professeure des Universités, section CNU64 - Université de Montpellier - France
-
2015-2023Maître de Conférences, section CNU64 - Université de Montpellier - France
-
2012-2015Postdoctorat - Prof. Dame C.V. Robinson' group - Université d’Oxford - Royaume-Uni
-
2009-2012Thèse - Laboratoire des Biomolécules UMR7203 - Paris - France
-
2023Prix de la Recherche, Société Française de Spectrométrie de Masse
-
2023-Élue représentante des enseignants-chercheurs de l’axe "Biologie Quantitative", Pôle Biologie-Santé, Université de Montpellier
-
2021-Co-responsable du parcours "Quantitative Biology", Master Biologie-Santé - Université de Montpellier
-
2020-2025Membre Junior de l’Institut Universitaire de France
-
2019-Responsable applications Spectrométrie de Masse structurale, FPP, PPM
-
2009-2012Bourse de thèse "Ministère de l’enseignement supérieur et de la recherche"
- Je suis biochimiste spécialisée dans l'application des approches de spectrométrie de masse structurale pour étudier la structure, dynamique et interactions des protéines.
- Mes études visent à caractériser la structure et la dynamique des récepteurs couplés aux protéines G, principale cible de médicaments, ainsi que leurs partenaires d’interaction. Pour ce faire, je combine les outils innovants de spectrométrie de masse structurale avec d’autres approches biophysiques et biochimiques plus "classiques".
- Mon projet principal vise à déchiffrer le rôle des GPCRs dans les interactions hôte/pathogène en utilisant la spectrométrie de masse native, l’échange hydrogène/deutérium, la pharmacologie ainsi que la biologie structurale. Cette étude permettra d'approfondir notre connaissance de ces récepteurs, tout en nous proposant une stratégie thérapeutique alternative pour lutter contre la résistance antimicrobienne.
- De plus, je collabore avec différentes équipes nationales et internationales pour analyser diverses protéines cibles par spectrométrie de masse structurale, afin de déterminer et caractériser leurs stœchiométries, modifications conformationnelles et partenaires d’interaction.

 IGF Nord 224
IGF Nord 224 04 34 35 92 80
04 34 35 92 80

 IGF Nord 204b
IGF Nord 204b 04 34 35 92 80
04 34 35 92 80-
2022HDR - Biologie & Santé - Université de Montpellier - France
-
2013Doctorat - Génomique Fonctionnelle et Structurelle - Scuola Internazionale Superiore di Studi Avanzati (SISSA) - Italie
-
2009Master - Biophysique - Beijing University of Technology - Chine
-
2005Licence - Chimie - Nankai University - Chine
-
2021-Postdoctorante - Institut de Génomique Fonctionnelle - Montpellier - France
-
2016-2020Postdoctorante - Université Côte d'Azur - Nice - France
-
2013-2016Postdoctorante - Forschungszentrum Jülich - Germany
-
2009-2013Doctorat - Scuola Internazionale Superiore di Studi Avanzati (SISSA) - Italie
-
2006-2009Master - Biophysique - Beijing University of Technology - Chine
-
2005-2006Coordinatrice du Marketing et des Ventes - Wendeng Mechanical Engineering Ltd. - Chine
- Distinctions
-
2019NeuroMod Bourse Postdoctorale - Université Côte d'Azur - France
-
2017Prix d'Excellence - Université Côte d'Azur - France
-
2016Bourse Postdoctorale - Fondation Allemande pour la Recherche (DFG) - Allemagne
-
2013Prix National Chinois pour les Meilleurs Doctorants à l’Étranger - Italie
- Responsabilités
-
2023-présent: Membre du Comité CT7 de GENCI - France
- Je suis biophysicienne/chimiste computationnelle, spécialisée dans les dynamiques structure-fonction des cibles de médicaments et des interactions protéine-médicament. Mes recherches intègrent la biologie structurale, la bio-/chimio-informatique, la biophysique et la pharmacologie, afin de mieux comprendre les mécanismes moléculaires des maladies et de l'action des médicaments.
- Principales techniques maitrisées
- - Simulations moléculaires : RCPGs et protéines transmembranaires
- - Conception de médicaments assistée par ordinateur et criblage virtuel
- - Apprentissage automatique
- - Exploration et analyse de données

 IGF Sud 025
IGF Sud 025 04 34 35 93 45
04 34 35 93 45-
2011Habilitation à Diriger les Recherches (HDR) - Université de Montpellier - France
-
1991Doctorat - Université de Montpellier - France
-
1991PhD - Université de Sherbrooke - Canada
-
1986Master 2 Biologie-Santé - Université de Montpellier - France
-
2011-Directeur de recherche - CNRS - Institut de Génomique Fonctionnelle (IGF) - Montpellier - France
-
1992-2010Chargé de recherche - CNRS - Institut de Génomique Fonctionnelle (IGF) - Montpellier - France
-
1991-1992Postdoctorant - Centre CNRS-INSERM de Pharmacologie-Endocrinologie (CCIPE) - Montpellier - France
-
1987-1990Doctorant - Université de Sherbrooke - Canada
-
2021-Directeur Adjoint - Institut de Génomique Fonctionnelle (IGF) - Montpellier - France
-
1988Prix Jean-Pierre Caillet - Université de Sherbrooke - Canada
- Électrophysiologiste de formation, je suis aujourd’hui spécialisé en pharmacologie moléculaire. Mes modèles d’étude sont les récepteurs couplés aux protéines G, en particulier les récepteurs de la vasopressine, des chimiokines et de la dopamine, avec pour question principale, l’étude de l’impact de l’oligomérisation des récepteurs sur leurs propriétés pharmacologiques.

 IGF Nord 226
IGF Nord 226 04 34 35 93 02
04 34 35 93 02-
2018Ph.D. en Biochimie et Médecine Moléculaire, option Dynamique cellulaire des complexes macromoléculaires - Université de Montréal - Montréal - Canada
-
2012Master Recherche en Bioinformatique, Biochimie Structurale et Génomique, option Biochimie Structurale - Faculté des sciences de Luminy - Marseille - France
-
2010Licence en Biochimie, Biologie, Terre et Environnement, option Biochimie - UFR Sciences Exactes - Reims - France
-
2023-Postdoctorant (Marie Curie MSCA) en Biologie Structurale - Institut de Génomique Fonctionnelle - Montpellier - France
-
2020-2023Postdoctorant en Biologie Structurale (CNRS et INSERM) - Institut de Génomique Fonctionnelle & Centre de Biologie Structurale - Montpellier - France
-
2019-2020Assistant de recherche (niveau R2) - Université de Montréal - Montréal - Canada
-
2018-2019Postdoctorant en Biochimie - Université de Montréal - Montréal - Canada
- Bourses d’études
-
2022Horizon Europe - Marie Curie grant
-
2022Bourse cryo-EM acquisition ESRF
-
2017Bourse de fin d’étude de la FESP
-
2016Bourse d’excellence de la FESP
-
2016Bourse d’excellence du CDMC
-
2015Bourse d’excellence de la FESP
-
2014Bourse d’excellence de la FESP
-
2014Bourse de competition du CDMC de 3 ans
- Prix/distinctions
-
2022Meilleure présentation orale YISS 2022, Institut de Génomique Fonctionnelle, Montpellier, France
-
2021"Seal of Excellence" - Horizon 2020-MSCA
-
2018Travel award de la faculté dentaire, Université de Montréal, Montréal, Canada
-
2018Travel award du RSBO, Montréal, Canada
-
2017Meilleure présentation orale, GRASP scientific day 2017, McGill university, Montréal, Canada
-
2017Travel award de la faculté dentaire, Université de Montréal, Montréal, Canada
-
2017Travel award du congrès CMSC, Montréal, Canada
-
2016Meilleure présentation orale, GRASP scientific day 2016, McGill university, Montréal, Canada
-
2016Travel award de la faculté dentaire, Université de Montréal, Montréal, Canada
-
2015Meilleure présentation poster, Proteo day 2015, Laval université, Québec, Canada
-
2015Meilleure présentation orale, Scientific day 2015 of Dental Medicine, Université de Montréal, Montréal, Canada
-
2014Travel award de la faculté dentaire, Université de Montréal, Montréal, Canada
- Missions scientifiques
-
2022-Membre nommé du Conseil de Laboratoire de l’IGF, Institut de Génomique Fonctionnelle, Montpellier
-
2021-Membre post-doctoral du comité BioSanté, partie structure, Montpellier
-
2022Organisation de la journée scientifique des PhD-PostDoc du Centre de Biologie Structurale, Montpellier
-
2021Organisation de la journée scientifique des PhD-PostDoc du Centre de Biologie Structurale, Montpellier
-
2021Organisation hebdomadaire des séminaires PhD-PostDoc, Centre de Biologie Structurale, Montpellier
-
2020-2022Membre du Conseil de Laboratoire du Centre de Biologie Structurale, Montpellier
-
2020Jury des présentations posters et orales de la journée de recherche dentaire 2020, Université de Montréal, Canada
-
2018-2020Membre de "International Association of Dental Research"
-
2017Animation scientifique ImageJ pendant le congrès CMSC, Montréal, Canada
-
2016Animation scientifique "La fête de la Science", Parc des expositions de Montréal, Montréal, Canada
-
2015Animation scientifique "La fête de la Science", Parc des expositions de Montréal, Montréal, Canada
- Biologie Structurale et imagerie : Transmission electron microscopy (single particle, tomography and tissue), Scanning electron microscopy (SEM), Focus-ion-beam-SEM, Atomic Force Microscopy, X-ray crystallography, Dichroisme circulaire, Microscopie à fluorescence, Microscopie à super-resolution (3DSIM, PALM/STORM).
- Biochimie : Méthodes de purification, Chromatographies d’affinité et d’exclusion de taille, SDS-gels, western blot, FACS, Immunofluorescence, préparation EM, Colloïdal gold immunolabeling.
- Biologie moléculaire : Clonage, purification d’ADN, PCR, qPCR, extraction et purification d’ARN, phage display.
- Biologie cellulaire : Culture cellulaire de cellules de mammifère et d’insectes, infection virale, Production de protéine, Détermination de modification post-traductionelle, test MTT, Essai Alamar blue.
- Biologie bactérienne : Culture bactérienne de bactéries aérobies et anaérobies, transformation bactérienne, test bacterial two-hybrid.
- Logiciels : Microsoft office, Sequencing software (primer3, Snapgene), RNAseq software (GSEA, pantherDB), EM analysis (Dragonfly, etomo, scipion, cryoSPARC, Relion), et logiciel de données d’analyse (ImageJ, Prism9, Pymol, CD)

 IGF Nord 213
IGF Nord 213 04 34 35 92 80
04 34 35 92 80

 IGF Nord 224
IGF Nord 224 04 34 35 93 20
04 34 35 93 20

 CHU
CHU 04 34 35 92 64
04 34 35 92 64

 IGF Nord 204b
IGF Nord 204b 04 34 35 92 80
04 34 35 92 80-
2014Qualification Maître de conférence (31 et 64)
-
2009Qualification Maître de conférence (28, 31 et 64)
-
2008Doctorat - Physique - Université de Grenoble Alpes - France
-
2005Ecole d’été "Protein NMR, recording, structure and evaluation" - Copenhague - Danemark
-
2004Master 2 - Physique des Sciences du Vivant - Université de Grenoble Alpes - France
-
2003Master 1 - Chimie-Physique - Université de Grenoble Alpes - France
-
2002Licence - Chimie-Physique - Université de Grenoble Alpes - France
-
2023-2024Chercheur Invité - Karolinska Institutet - Stockholm - Suède
-
2017-Chargé de Recherche INSERM - Institut de Génomique Fonctionnelle - Montpellier - France
-
2014-2017Postdoctorat - Institut de Génomique Fonctionnelle - Montpellier - France
-
2013-2014Postdoctorat - Institut de Biologie Structurale - Grenoble - France
-
2008-2013Postdoctorat - Harvard Medical School - Boston - MA - USA
-
2004-2008Thèse - Institut de Biologie Structurale - Grenoble - France
- Missions scientifiques
-
2023-Président du Comité Utilisateurs FRISBI
-
2019-2022Membre du Comité Utilisateurs FRISBI
-
2015-2017Membre du Conseil de Laboratoire (nommé) - Institut de Génomique Fonctionnelle
- Prix/Distinctions
-
2016Prix des Grandes Avancée en Biologie-Santé du Pôle Rabelais à Montpellier
-
2016Mention Spéciale au Prix Sabatier d’Espeyran de l’Académie des Sciences et des lettres de Montpellier
-
2015Prix Instruct "Research and Developement Pilot Project"
-
2014Prix du meilleur poster à la 3ème GDR3545.
-
2007Bourse pour la participation à la 48ème conférence ENC.
-
2006Bourse pour la participation à la 1ère conférence EUROMAR.
-
2005Prix du meilleur poster à la VIIIème Rencontre Rhône-Alpes de RMN.
-
2005Bourse pour l’école d’été "Protein NMR, Recording, Structure and Evaluation"
-
2004Bourse ministérielle (MENRT)
-
2003Bourse d’excellence Master 2 de l’UJF
- Au cours de mon doctorat à l'Institut de Biologie Structurale (IBS Grenoble, 2004-2007), et de ma recherche postdoctorale à Harvard Medical school (Boston -MA- USA, 2008-2013), j'ai acquis une expertise en spectroscopie RMN et développé des compétences importantes dans le marquage isotopique spécifique et les études structurales des protéines membranaires, ce qui m'a permis d'étudier les changements conformationnels des protéines transmembranaires.
- Cette expertise m’a permis de rejoindre l'Institut de Génomique Fonctionnelle (IGF Montpellier, 2014-2017) avant d’être recruté comme Chargé de Recherche par l’INSERM pour étudier les mécanismes moléculaires et structuraux de l'activité des récepteurs membranaires et de leurs partenaires, en particulier les récepteurs opioïdes et les récepteurs frizzled.

 IGF Nord 226a
IGF Nord 226a 04 34 35 93 02
04 34 35 93 02-
1990Maitrise de Biologie cellulaire - Université Paul Sabatier - Toulouse - France
-
1991DU Informatique de Gestion et Contrôle des Processus - Université Montpellier II - Montpellier - France
-
2014-Ingénieur d’Étude (IE) CNRS en Techniques Biologiques - IGF - Montpellier - France
-
2003-2013Assistant Ingénieur (AI) CNRS en Techniques Biologiques - IGF - Montpellier - France
-
2011-Conseillère en Radioprotection pour l'IGF
- Culture cellulaire
- Pharmacologie (FRET : binding, mesure production de seconds messagers)
- Biochimie : purification de RCPG
- Biologie moléculaire
- Encadrement des étudiants
- Lab manager : gestion des stocks, commandes sur Geslab, entretien des appareillages
- Analyse de données :
- GraphPad Prism
- Maîtrise du Pack office

 IGF Nord 207c
IGF Nord 207c 04 34 35 92 64
04 34 35 92 64

 IGF Sud 213
IGF Sud 213 04 34 35 92 80
04 34 35 92 80

 IGF Nord 207c
IGF Nord 207c 04 34 35 92 64
04 34 35 92 64

 IGF Nord 204a
IGF Nord 204a 04 34 35 92 80
04 34 35 92 80-
2017Doctorat de Biochimie, Biologie moléculaire et Cellulaire - Collège de France - Université Paris 5 Descartes - France
-
2013Master de Biologie Moléculaire et Cellulaire - Université Pierre et Marie Curie Paris 6 (UPMC) - France
-
2011Licence Sciences et Technologies- Université Pierre et Marie Curie Paris 6 (UPMC) - France
-
2021-2022Postdoctorat, équipe Dr. D. Gillet, Institut Joliot - Commissariat à l'énergie atomique et aux énergies alternatives (CEA) - France
-
2017-2020Postdoctorat, équipe Pr. M. Bouvier, Unité de recherche en pharmacologie moléculaire, Institut de Recherche en Immunologie et en Cancérologie - Université de Montréal - Canada
-
2013-2017Doctorat de Sciences en Biochimie, équipe du Dr. C. Llorens-Cortes, Centre Interdisciplinaire de Recherche en Biologie - Collège de France-Université Paris 5 Descartes - France
-
2012-2013Stage de recherche de M2, équipe du Dr. C. Llorens-Cortes, Centre Interdisciplinaire de Recherche en Biologie - Collège de France - Paris - France
-
2012Stage de recherche de M1, équipe du Dr. A. Couvineau, Centre de Recherche Biomédicale Bichat Beaujon - Paris - France
-
2011Stage de recherche, équipe du Dr. A. Couvineau, Centre de Recherche Biomédicale Bichat Beaujon - Paris - France
-
2008Stage de recherche dans l’équipe du Pr. Daniel Tomé, UMR 914 Physiologie de la nutrition et du comportement alimentaire - Paris - France
-
2017Bourse d’échange postdoctorale INSERM/FRQS #264714
-
2016Bourse d’Excellence Labex Memolife, financement 4ème année de thèse, concours interne Cirb
- Je suis un chercheur postdoctoral en pharmacologie, biochimie, biologie moléculaire et cellulaire. Je suis spécialisé dans l’étude des récepteurs couplés aux protéines G ainsi que dans le développement de biosenseurs de Resonance Energy Transfer permettant l’étude des interactions protéines/protéines et de leurs trafic intracellulaires.
- Mes différentes compétences :
- Biologie moléculaire : Clonage, PCR, mutagenèse dirigée, préparation d’ADN, développement et utilisation de biosenseurs d’interaction protéine-protéine ou de changements conformationnels intramoléculaires utilisant le Bioluminescence Resonnance Energy Transfer (BRET), Mesure de la production de seconds messagers (AMPc, IP3) par Förster Resonance Energy Transfer (FRET)
- Biologie Cellulaire : Culture cellulaire, établissement de lignées stables, préparation de membranes de cellules, microscopie confocale à fluorescence, marquage immunofluorescence
- Biochimie : Purification de protéines membranaires, caractérisation enzymologique (paramètres cinétiques, profils d’inhibition), Western Blot, ELISA. Biodistribution de molécules radiomarquées dans broyats d’organes
- Immunohistochimie : Coupes histologiques de différents organes en paraffine ou OCT et immunomarquages

 IGF Nord 204b
IGF Nord 204b 04 34 35 92 80
04 34 35 92 80

 IGF Nord 204b
IGF Nord 204b 04 34 35 92 80
04 34 35 92 80

 IGF Nord 207b
IGF Nord 207b 04 34 35 92 64
04 34 35 92 64

 IGF Nord 204b
IGF Nord 204b 04 34 35 92 80
04 34 35 92 80

 IGF Nord 204b
IGF Nord 204b 04 34 35 92 80
04 34 35 92 80

 IGF Nord 204a
IGF Nord 204a 04 34 35 92 80
04 34 35 92 80

 IGF Nord 207b
IGF Nord 207b 04 34 35 92 64
04 34 35 92 64

 IGF Nord 213
IGF Nord 213 04 34 35 92 80
04 34 35 92 80

 IGF Nord 213
IGF Nord 213 04 34 35 92 80
04 34 35 92 80
- Bous J, Fouillen A, Orcel H, Trapani S, Cong X, Fontanel S, Saint-Paul J, Lai-Kee-Him J, Urbach S, Sibille N, Sounier R, Granier S#, Mouillac B#, Bron P#. Structure of the vasopressin hormone-V2 receptor-β-arrestin1 ternary complex. Science Adv. 2022 Sep 2;8(35):eabo7761. doi: 10.1126/sciadv.abo7761. # Co-corresponding authors.
- Healey RD, Saied EM, Cong X, Karsai G, Gabellier L, Saint Paul J, Del Nero E, Jeannot S, Drapeau M, Fontanel S, Maurel D, Basu S, Leyrat C, Golebiowski J, Bossis G, Bechara C, Hornemann T, Arenz C, Granier S. Discovery and mechanism of action of small molecule inhibitors of ceramidases. Angew Chem Int Ed Engl. 2021 Oct 20. doi: 10.1002/anie.202109967.
- Grison CM, Lambey P, Jeannot S, Del Nero E, Fontanel S, Peysson F, Heuninck J, Sounier R, Durroux T, Leyrat C, Granier S#, Bechara C#. Molecular insights into mechanisms of GPCR hijacking by Staphylococcus aureus. Proc Natl Acad Sci U S A. 2021 Oct 19;118(42):e2108856118. doi: 10.1073/pnas.2108856118. # Co-corresponding authors.
- Cong X, Maurel D, Déméné H, Vasiliauskaité-Brooks I, Hagelberger J, Peysson F, Saint-Paul J, Golebiowski J, Granier S#, Sounier R#. Molecular insights into the biased signaling mechanism of the μ-opioid receptor. Mol Cell. 2021 Oct 21;81(20):4165-4175.e6. doi: 10.1016/j.molcel.2021.07.033. Epub 2021 Aug 24. # Co-corresponding authors.
- Bous J, Orcel H, Floquet N, Leyrat C, Lai-Kee-Him J, Gaibelet G, Ancelin A, Saint-Paul J, Trapani S, Louet M, Sounier R, Déméné H, Granier S#, Bron P#, Mouillac B#. Cryo-electron microscopy structure of the antidiuretic hormone arginine-vasopressin V2 receptor signaling complex. Sci Adv. 2021 May 21;7(21). doi: 10.1126/sciadv.abg5628. # Co-corresponding authors.
- Vasiliauskaité-Brooks I, Healey RD, Rochaix P, Saint-Paul J, Sounier R, Grison C, Waltrich-Augusto T, Fortier M, Hoh F, Saied EM, Arenz C, Basu S, Leyrat C, Granier S#. Structure of a human intramembrane ceramidase explains enzymatic dysfunction found in leukodystrophy.Nature Commun. 2018 Dec 21;9(1):5437.
- Vasiliauskaité-Brooks I, Sounier R, Rochaix P, Bellot G, Fortier M, Hoh F, De Colibus L, Bechara C, Saied EM, Arenz C, Leyrat C#, Granier S#. Structural insights into adiponectin receptors suggest ceramidase activity. 2017 Apr 06;544:120-123. doi: 10.1038/nature21714. # Co-corresponding authors.
- Sounier R, Mas C, Steyaert J, Laeremans T, Manglik A, Huang W, Kobilka BK, Déméné H, Granier S. Propagation of conformational changes during μ-opioid receptor activation. Nature. 2015 Aug 20;524(7565):375-8.
- Granier S, Manglik A, Kruse AC, Kobilka TS, Thian FS, Weis WI, Kobilka BK. Structure of the δ-opioid receptor bound to naltrindole. 2012 May 16;485(7398):400-4.
- Manglik A, Kruse AC, Kobilka TS, Thian FS, Mathiesen JM, Sunahara RK, Pardo L, Weis WI, Kobilka BK, Granier S. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. 2012 Mar 21;485(7398):321-6.
Bases moléculaires et structurales de la fonction des GPCRs
Responsables
Rémy SOUNIER, Thierry DURROUX et Christiane MENDRE