PLAGL1/ZAC1 RÉGULE LE RÉSEAU DES GÈNES SOUMIS À EMPREINTE ET LES GÈNES DE LA MATRICE EXTRACELLULAIRE
L’IDENTIFICATION DES SITES DE LIAISON ET DES GÈNES CIBLES DE PLAGL1/ZAC1 DÉMONTRE SON RÔLE DANS LA RÉGULATION DES GÈNES DE LA MATRICE EXTRACELLULAIRE ET DU RÉSEAU DES GÈNES SOUMIS À EMPREINTE.
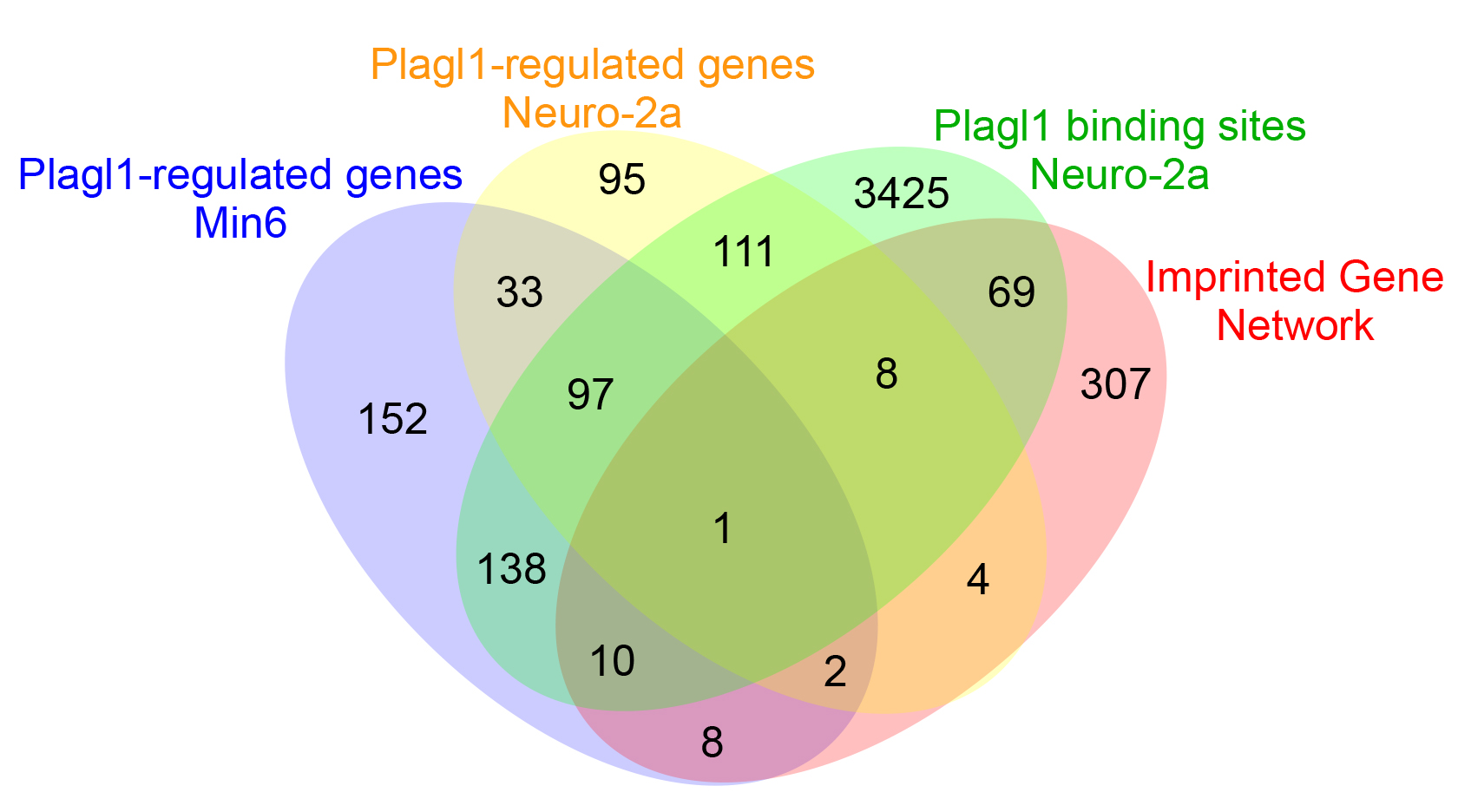
PLAGL1/ZAC1 est un gène soumis à empreinte génomique parentale d’expression paternelle qui fait partie du réseau des gènes soumis à empreinte. Il code un facteur de transcription à doigts de zinc montrant une activité antiproliférative et est un gène suppresseur de tumeur candidat situé en 6q24 dont l'expression est fréquemment perdue dans divers néoplasmes. À l'inverse, un gain de fonction de PLAGL1 est responsable du diabète néonatal transitoire, une maladie génétique rare qui résulte du développement anormal du pancréas. Dans le présent travail, nous avons montré que l’induction de Plagl1 n'était pas associée à l'arrêt du cycle cellulaire induit par les dommages à l'ADN. Il était plutôt associé à des arrêts physiologiques du cycle cellulaire tels que l'inhibition de contact, la déprivation en facteurs de croissance ou la différenciation cellulaire. Pour mieux comprendre le mécanisme d'action de Plagl1, nous avons identifié ses gènes cibles en combinant immuno-précipitation de la chromatine (ChIP) et transcriptomique (DGE, RNA-seq) dans des lignées cellulaires transfectées. L’induction des gènes cibles de Plagl1 est corrélée à une liaison multiple à des motifs riches en GC de la région promotrice proximale. Les gènes cibles de Plagl1 incluent de nombreux gènes impliqués dans la signalisation, l'adhésion cellulaire et la composition de la matrice extracellulaire, y compris les collagènes. Les cibles de Plagl1 comprennent également 22% des 409 gènes qui forment l'IGN. En résumé, ce travail montre que Plagl1 code un facteur de transcription qui coordonne la régulation d'un sous-ensemble des gènes du réseau des gènes soumis à empreinte et module la composition de la matrice extracellulaire.
Analyse de l’enrichissement des termes Gene Ontology (GO) et des voies KEGG parmi les gènes cibles de Plagl1. Les gènes régulés par Plagl1 dans les neuroblastomes murins Neuro-2a ont été analysés en utilisant les outils disponibles sur le site DAVID (https://david.ncifcrf.gov/).
L’illustration montre le facteur d’enrichissement de chaque terme GO ou voie KEGG. La couleur des points indique la catégorie des termes GO ou des voies KEGG. Leur taille est proportionnelle au nombre de gènes concernés.
Les carrés colorés à proximité de chaque terme GO ou voie KEGG indiquent la valeur de la probabilité d’observation, ajustée selon la méthode de Benjamini-Hochberg.
Les différents termes GO et voies KEGG ont été groupés selon leur similarité sémantique.
Lien PUBLICATION
Contact : ; ; +33 (0)4 34 35 92 41
IDENTIFICATION OF PLAGL1/ZAC1 BINDING SITES AND TARGET GENES ESTABLISHES ITS ROLE IN THE REGULATION OF EXTRACELLULAR MATRIX GENES AND THE IMPRINTED GENE NETWORK
PLAGL1/ZAC1 undergoes parental genomic imprinting, is paternally expressed, and is a member of the imprinted gene network (IGN). It encodes a zinc finger transcription factor with anti-proliferative activity and is a candidate tumor suppressor gene on 6q24 whose expression is frequently lost in various neoplasms. Conversely, gain of PLAGL1 function is responsible for transient neonatal diabetes mellitus, a rare genetic disease that results from defective pancreas development. In the present work, we showed that Plagl1 up-regulation was not associated with DNA damage-induced cell cycle arrest. It was rather associated with physiological cell cycle exit that occurred with contact inhibition, growth factor withdrawal, or cell differentiation. To gain insights into Plagl1 mechanism of action, we identified Plagl1 target genes by combining chromatin immuno-precipitation and genome-wide transcriptomics in transfected cell lines. Plagl1-elicited gene regulation correlated with multiple binding to the proximal promoter region through a GC-rich motif. Plagl1 target genes included numerous genes involved in signaling, cell adhesion, and extracellular matrix composition, including collagens. Plagl1 targets also included 22% of the 409 genes that make up the IGN. Altogether, this work identified Plagl1 as a transcription factor that coordinated the regulation of a subset of IGN genes and controlled extracellular matrix composition.
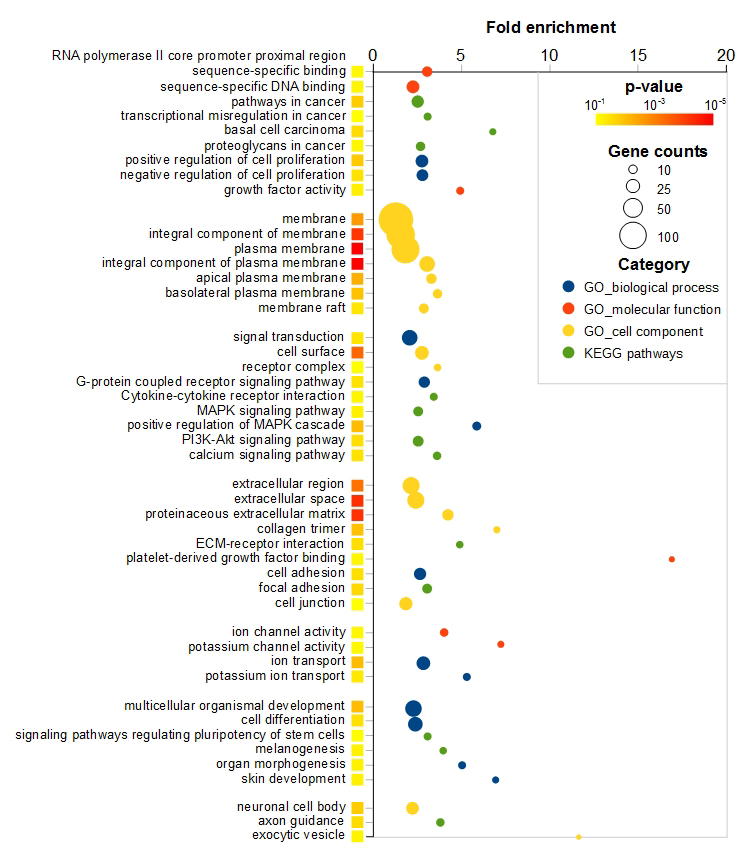 GO terms and KEGG pathways enrichment analysis. Genes regulated by Plagl1 transfection into Neuro-2a cells were analyzed using the DAVID bioinformatics resources (https://david.ncifcrf.gov/).
GO terms and KEGG pathways enrichment analysis. Genes regulated by Plagl1 transfection into Neuro-2a cells were analyzed using the DAVID bioinformatics resources (https://david.ncifcrf.gov/).
The figure displays the fold-enrichment of each statistically significant GO term/KEGG pathway. Circle color maps the GO term/KEGG pathway category.
Circle size maps the number of genes in each GO term/KEGG pathway. The colored square next to each GO term/KEGG pathway maps the Benjamin-Hochberg-corrected p-value.
The different GO terms/KEGG pathways were grouped according to their semantic similarities.
Publication link
Contact : ; ; +33 (0)4 34 35 92 41





