TOPOFUN : QUAND L’APPRENTISSAGE AUTOMATISÉ PERMET DE RECONNAITRE DES GÈNES FONCTIONNELLEMENT LIÉS
Attribuer au moins une fonction à chaque gène et identifier les gènes qui concourent à une même fonction sont deux des buts fondamentaux de la génomique fonctionnelle. La co-expression fréquente d’un ensemble de gènes est l’indice que ces gènes sont fonctionnellement liés, c’est-à-dire qu’ils participent à au moins un même processus biologique. En accord avec la définition classique d’un module fonctionnel par Hartwell et al. (From molecular to modular cell biology. Nature, 1999, 402, C47–52), les méthodes permettant d’identifier de tels modules fonctionnels dans des réseaux de co-expression génique font toutes l’hypothèse que les membres d’un module ont plus de relations entre eux qu’avec les membres d’autres modules. Cette hypothèse est vérifiable dans des réseaux comprenant peu de gènes et l’identification de modules fonctionnels est relativement aisée. Dès que la taille des réseaux étudiés augmente, cette hypothèse ne peut pas être facilement testée. Nous avons donc choisi de ne pas faire d’hypothèse sur la topologie des modules fonctionnels mais plutôt d’apprendre leur topologie à partir de modules fonctionnels connus. Cette connaissance peut ensuite être utilisée pour identifier de nouveaux modules fonctionnels, trouver de nouveaux membres de modules déjà identifiés ou identifier les gènes fonctionnellement liés à un gène donné.
Les modules fonctionnels (MF) connus que nous avons utilisés sont les 978 ‘’Processus Biologiques’’ (PB) comprenant entre 20 et 500 gènes, tels que définis dans la base de données Gene Ontology (GO). Nous avons également constitué un nombre équivalent de modules aléatoires (MA) de même taille par tirage au hasard dans l’ensemble des gènes du réseau. Nous avons ensuite calculé les valeurs de 12 paramètres caractérisant la topologie des modules étudiés : degré, connectivité du voisinage, plus court chemin, coefficient de clustering, centralité de proximité... Nous avons utilisé la technique LASSO (Least Absolute Shrinkage and Selection Operator) pour identifier six paramètres qui différenciaient au mieux MA et MF et avons réalisé une analyse discriminante linéaire pour trouver une combinaison linéaire de ces paramètres. Cette procédure a permis d’attribuer un score topologique (ScoreTopo) à chaque module. Ce score est élevé si la topologie du module ressemble à celle d’un PB connu et faible si elle ressemble à celle d’un MA. Parallèlement, nous avons utilisé la méthode de Wang (Wang JZ et al. (2007) A new method to measure the semantic similarity of GO terms. Bioinformatics, 23, 1274–81.) pour définir un score fonctionnel (ScoreFun) pour chaque module. Ce score est basé sur la distance des annotations des gènes du module dans l’arbre reliant les termes GO. Ce score est élevé si les gènes qui constituent le module sont fonctionnellement proches et faible dans le cas contraire. La combinaison des 2 scores permet de mesurer à quel point un module candidat est constitué de gènes aussi fonctionnellement apparentés que ceux des PB GO.
Le nombre de modules candidats dans un réseau d’environ 20.000 gènes est beaucoup trop grand pour envisager une exploration exhaustive ; nous avons choisi d’utiliser un algorithme génétique que nous avons nommé TopoFun. ScoreTopo et ScoreFun ont été combinés dans une fonction d’évaluation (fitness function) capable de classer différents modules. En partant d’une population de modules candidats, en les faisant évoluer par mutation (mutation ponctuelle, insertion, délétion, recombinaison) et en les sélectionnant grâce à la fonction d’évaluation, l’algorithme converge après quelques milliers d’itérations vers un module qui représente une excellente solution (à défaut d’avoir la certitude qu’il s’agit de la meilleure).
Afin de confirmer l’intérêt de notre méthode, nous sommes partis de PB connus et avons essayé de les ‘’améliorer’’ en cherchant dans le réseau de co-expression de nouveaux gènes, non annotés par ce PB, mais dont la co-expression avec les membres connus de ce PB suggère qu’ils concourent à la même fonction. En utilisant une base de données indépendante (STRING), nous avons vérifié que les nouveaux gènes ainsi obtenus étaient significativement liés fonctionnellement à ceux déjà connus. Enfin, nous avons évalué les modules trouvés par WGCNA (Weighted Gene Co-expression Network Analysis), la méthode de référence en analyse de réseaux de co-expression. Ces modules ont une excellente topologie, ‘’supérieure’’ à celles des PB connus. Ils souffrent cependant d’une faible cohérence fonctionnelle et ressemblent plus à des MA de ce point de vue. Nous avons donc utilisé TopoFun pour les améliorer et avons produit des modules présentant une topologie très légèrement améliorée mais surtout une cohérence fonctionnelle bien supérieure.
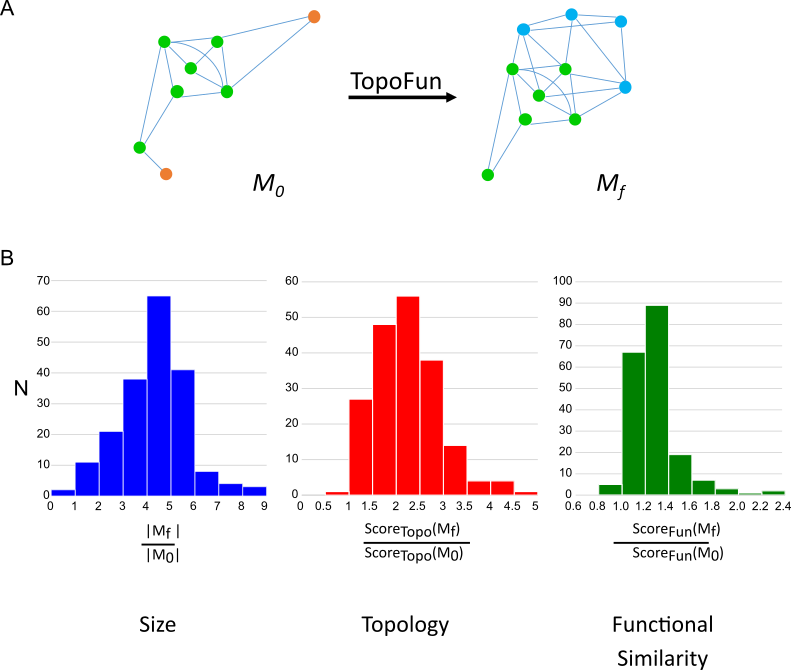
En partant d’un ensemble de gènes donnés (A, M0), TopoFun élimine les gènes les moins co-exprimés (orange)
et identifie de nouveaux gènes (bleu) co-exprimés avec ceux du module original, tout en améliorant (B) la taille, la topologie et la similarité sémantique du module final (Mf).
Lien publication
TopoFun: a machine learning method to improve the functional similarity of gene co-expression modules. Janbain A, Reynès C, Assaghir Z, Zeineddine H, Sabatier R, Journot L. NAR Genom Bioinform, 2021.
.....................................................................................................................................................................................................................................................................................................................................................................
TOPOFUN, A MACHINE LEARNING-BASED METHOD TO IDENTIFY FUNCTIONALLY RELATED GENES
The functional annotation of each gene and the identification of genes involved in each function are two basic goals of functional genomics. The frequent co-expression of members of a gene set is an indication that the constituting genes are functionally related and participate to one or more common biological process(es). Following the classical definition of functional modules by Hartwell and colleagues (From molecular to modular cell biology. Nature, 1999, 402, C47–52), the methods aimed at identifying functional modules all assume that the members of a module have more links with one another than they have with genes in other modules. This hypothesis can be verified in networks comprising a limited number of genes and the identification of functional modules is then relatively easy. When the number of genes increases, this hypothesis is more difficult to test. We decided to make no hypothesis about the functional modules topology; we rather learned their topology from known functional modules using machine learning. This knowledge is then useful to identify new functional modules, to find new members of known functional modules, and to identify functionally related genes for a given seed gene.
We studied the 978 Gene Ontology Biological Processes (GO-BPs) with 20-500 genes as prototypic functional modules (FMs). We generated an equivalent number of random modules (RMs) made of equivalent numbers of randomly sampled genes. We computed 12 topological descriptors of the modules topology: degree, neighborhood connectivity, shortest path, clustering coefficient, closeness centrality... Using LASSO (Least Absolute Shrinkage and Selection Operator), we selected the six topological descriptors that best discriminated FMs and RMs. Using the selected topological descriptors, we performed LDA (Linear Discriminant Analysis) to construct a topological score (ScoreTopo) that predicted the type of a module, random-like or functional-like. Based on the work of Wang and colleagues (A new method to measure the semantic similarity of GO terms. Bioinformatics, 2007, 23, 1274–81), we designed a functional similarity score (ScoreFun) based on the distance in the GO tree of the annotations of the genes that constitute a module; the score is high if the module’s genes are functionally close and low otherwise. ScoreTopo and ScoreFun estimate to which extent a module is made of genes as functionally related as those of GO-BPs.
The number of candidate functional modules in a network of about 20,000 genes is much too high for a systematic assessment. We combined ScoreTopo and ScoreFun in a fitness function that ranked candidate modules and we designed a genetic algorithm we named TopoFun to explore the co-expression network. Starting from a population of candidate modules to which we applied mutations (point mutations, insertions, deletions, crossing-over) and selection using the fitness function, TopoFun converged after a few thousands of iterations towards a module representing a very good solution (although we cannot prove it is the best one).
To illustrate the use of TopoFun, we started from a subset of the Gene Ontology Biological Processes (GO-BPs) and showed that TopoFun efficiently retrieved genes that we omitted, and aggregated a number of novel genes to the initial GO BP while improving module topology and functional similarity. Using an independent protein-protein interaction database (STRING), we confirmed that the novel genes gathered by TopoFun were functionally related to the original gene set. Finally, we evaluated the functional modules produced by WGCNA (Weighted Gene Co-expression Network Analysis), the reference method in network algorithms. WGCNA modules display an exceptional topology, better than that of known GO-BPs. However, they lack functional coherence and are not significantly different from RMs from that point of view. We used TopoFun to improve the WGCNA modules functional similarity and produced modules with a slightly improved topology and much higher functional consistency.
Publication link
TopoFun: a machine learning method to improve the functional similarity of gene co-expression modules. Janbain A, Reynès C, Assaghir Z, Zeineddine H, Sabatier R, Journot L. NAR Genom Bioinform, 2021.